Científicos del Instituto Ciencias de Mar del CSIC, la Universidad de la Laguna y la Universidad de Alicante participan en un estudio sobre los genomas de las bacterias de la superficie marina que publica esta semana en PNAS. El trabajo, liderado por el Dr. Stepanauskas del Bigelow Laboratory for Ocean Sciences, en Maine, EE. UU., junto con otros laboratorios americanos, indica que el ADN de dichas bacterias contiene menos duplicidades, genes no codificantes, guanina y citosina que el de las bacterias cultivables.
Científicos del Instituto Ciencias de Mar del CSIC, la Universidad de la Laguna y la Universidad de Alicante participan en un estudio sobre los genomas de las bacterias de la superficie marina que publica esta semana en PNAS. El trabajo, liderado por el Dr. Stepanauskas del Bigelow Laboratory for Ocean Sciences, en Maine, EE. UU., junto con otros laboratorios americanos, indica que el ADN de dichas bacterias contiene menos duplicidades, genes no codificantes, guanina y citosina que el de las bacterias cultivables.
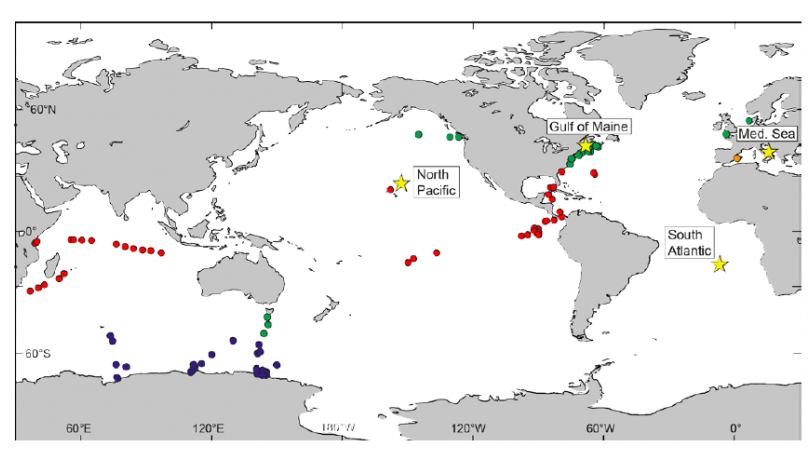
Las bacterias marinas tienen una función primordial en los ciclos biogeoquímicos del océano y son la base de la cadena trófica. Por ejemplo, la fijación de CO2 y la formación de O2 en los océanos suponen aproximadamente la mitad del balance global en la Tierra. Las algas y bacterias fotosintéticas (cianobacterias) llevan a cabo este proceso, mientras que las bacterias que no fijan CO2 consumen la materia orgánica que se genera en el océano. Sin embargo, hasta hace apenas 5 años, la única manera de tener la información completa de un genoma bacteriano era a través de su aislamiento en cultivo. Este método tiene el gran problema de que apenas un 1% de las bacterias que son realmente abundante en el mar se pueden cultivar en el laboratorio, lo que dificulta enormemente el conocimiento biológico de la gran mayoría de las bacterias del océano. Esta limitación ha sido superada gracias a la nuevas tecnologías de separación de células a nivel individual a partir de muestras ambientales y de secuenciación masiva. Esta tecnología que en inglés se llama “Single-Cell Genomics” (Genómica a partir de una única célula) se ha aplicado por primera vez de una forma integrada y se han analizado decenas de nuevos genomas marinos de bacterias ubicuas procedentes de diversos océanos del mundo, incluyendo el Océano Atlántico Norte y Sur, Pacífico Sur, y el Mar Mediterráneo según publica esta semana la revista Proceedings of the National Academy of Sciences (PNAS).
ADAPTACION A LAS CONDICIONES OLIGOTROFICAS DEL MAR
La investigadora del Instituto Ciencias del Mar del CSIC, Silvia G. Acinas explica: "La importancia de este estudio radica en que es la primera vez que se analizan los genomas marinos realmente abundantes y representativos de la capa fótica del mar (hasta los 200 m de profundidad) sin tener el sesgo del aislamiento en cultivo. Es sorprendente cómo los genomas bacterianos han reducido sus genomas y se han especializado metabólicamente de formas muy diversas a las condiciones que podrían ser adversas para el crecimiento de los microorganismos en los océanos".
“La simplificación del genoma supone un ahorro de energía al no contener este tanta información genética, dicha energía puede ser reinvertida en otros procesos esenciales de la célula” señala Acinas. De hecho, los genomas cultivados suelen ser hasta el doble de grandes que los de las bacterias de vida libre analizadas por la investigación.
Gracias a la secuenciación de estos genomas bacterianos marinos sabemos que tienen menos duplicaciones de genes, menos genes no codificantes, menor contenido en GC y han reducido su maquinaria de transcripción genética y de síntesis de proteínas al mínimo cuando se los compara con los microorganismos cultivables. Esto último ha tenido como consecuencia una reducción en la versatilidad metabólica pero, a la vez, ha permitido una gran especialización funcional. "La visión genómica y funcional que teníamos de las bacterias marinas estaba totalmente sesgada debido a la información previa que teníamos de los bacterias aisladas en cultivo" asegura el investigador de la Universidad de la Laguna José M. Gonzalez.
BIOGEOGRAFIA DE LAS BACTERIAS MARINAS
Gracias a este estudio también se ha podido analizar la distribución espacial de dichos genomas bacterianos en los distintos océanos del mundo. Debido a su ubiquidad y su abundancia, al tamaño microscópico de las bacterias y a la inexistencia de barreras geográficas que limiten su distribución espacial, se había pensado que las bacterias marinas no presentaban patrones biogeográficos porque pueden dispersarse globalmente. Según apunta el investigador de la Universidad de Alicante Manuel Martínez García " utilizando la información completa de los genomas bacterianos marinos de este estudio se ha observado una distribución clara de estos microorganismos que se correlaciona con la temperatura y la latitud, lo que pone de manifiesto la existencia de patrones biogeográficos en los microorganismos marinos y su adaptación a las condiciones medioambientales locales".
Como concluye Silvia G. Acinas, del ICM, “para tener un conocimiento real del papel que realizan las bacterias marinas, tan importantes en los ciclos biogeoquímicos del océano, es imprescindible conocer la secuencia de su genoma”.
Publicación:
Brandon K. Swan, Ben Tupper, Alexander Sczyrba, Federico M. Lauro, Manuel Martinez-Garcia, José M. González, Haiwei Luo, Jody J. Wright, Zachary C. Landry, Niels W. Hanson, Brian Thompson, Nicole J. Poulton, Patrick Schwientek, Silvia G. Acinas, Stephen J. Giovannoni, Mary Ann Moran, Steven J. Hallam, Ricardo Cavicchioli, Tanja Woyke and Ramunas Stepanauskas. 2013. Prevalent genome streamlining and latitudinal divergence of planktonic bacteria in the surface ocean.Proceedings of the National Academy of Sciences. DOI: 10.1073/pnas.1304246110